- Áreas de conocimiento: Química
- Unidad Académica: FACULTAD DE CIENCIAS EXACTAS
- Lugar de Trabajo: Laboratorio de Investigación y Desarrollo de Bioactivos (LIDeB)
- Beca: Doctoral
- Otorgada por: CONICET
- Contacto: juanialice185@gmail.com
Búsqueda de nuevos farmacos antichagásicos inhibidores de la tripanotión sintetasa
Juan Ignacio Alice
Director/a/e: Talevi Alan
Codirector/a/e: Bellera Carolina
Resumen
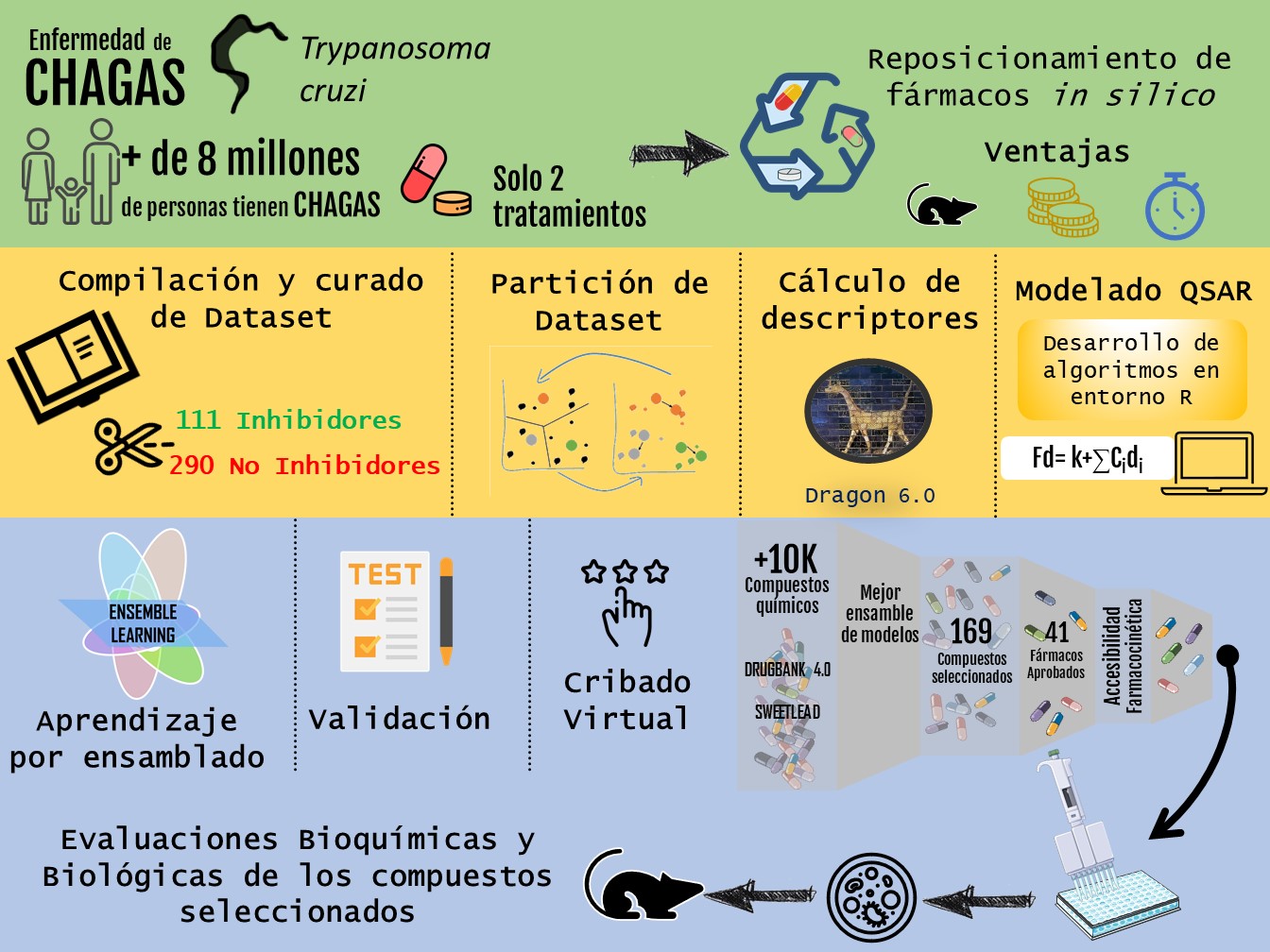
El plan de trabajo tiene como objetivo la búsqueda de nuevos tratamientos antichagásicos a partir de herramientas computacionales, focalizando dicha búsqueda en compuestos con posible actividad inhibitoria de la enzima Tripanotión Sintetasa (TryS). La Enfermedad de Chagas es una parasitosis endémica de América Latina causada por el protozoo hemoflagelado Tripanosoma cruzi (T. cruzi). Actualmente afecta a más de 8 millones de personas y genera más de 12.000 muertes anuales (https://www.who.int/health-topics/chagas-disease; último acceso 5/10/2020). A pesar de ello solo se disponen de 2 drogas para su tratamiento, las cuales resultan inadecuadas por su baja tasa de curación en la etapa crónica de la enfermedad, y por sus severos efectos adversos. Es por lo tanto necesario encontrar nuevas alternativas terapéuticas que puedan superar estas limitaciones. En nuestro trabajo, desarrollamos modelos in silico para seleccionar inhibidores de TryS, para ello empleamos un método de semicorrelación con el objetivo de obtener modelos clasificadores lineales, con capacidad de clasificar un compuesto como Activo (con capacidad inhibitoria sobre TryS) o Inactivo (sin efecto inhibitorio), basando el cálculo en descriptores moleculares. A partir de una búsqueda bibliográfica se construyó una base de datos con más 400 entidades químicas evaluadas frente a TryS. Mediante scripts in house en entorno R se generaron 1000 modelos capaces de discriminar entre compuestos con y sin capacidad inhibitoria. A fin de validar los modelos se llevó a cabo una campaña piloto de cribado in silico (cribado retrospectivo) sobre una biblioteca de compuestos conteniendo una pequeña proporción de inhibidores conocidos dispersos entre señuelos generados mediante la aplicación DUD-E (Mysinger et al., 2012). Los modelos con mejor desempeño fueron combinados mediante aprendizaje por ensamblado. Se utilizaron diversas métricas de enriquecimiento para evaluar el desempeño de los modelos, y mediante el análisis de gráficas de superficie del PPV (Morales et al., 2020) optimizamos el valor de corte del score de manera de disminuir considerablemente la probabilidad de encontrarnos con falsos positivos. El ensamble de los mejores modelos fue aplicado al cribado in silico de las bases de datos SweetLead (Novick et al., 2013) y DrugBank (Knox et al., 2011), seleccionando 169 compuestos químicos (incluyendo 41 fármacos aprobados) como potenciales compuestos tripanocidas. Finalmente, luego de un análisis de las características de la patología, estructura química y propiedades biofarmacéuticas de los candidatos seleccionados, adquirimos 5 drogas para evaluar su actividad inhibitoria frente enzima TryS de tres especies de tripanosomátidos (T. cruzi, Tripanosoma brucei (T. brucei) y Leishmania infantum) y actividad tripanocida frente a diferentes estadios de T. cruzi y al estadio sanguíneo de T. brucei. VER VIDEO
Palabras Claves: CHAGAS | DISEÑO DE FARMACOS | MACHINE LEARNING | QSAR | TRYPANOSOMA CRUZI
Keywords: CHAGAS | DRUG DISCOVERY | MACHIN LEARNING | QSAR | TRYPANOSOMA CRUZI
